-
人源化和全人源抗体的CDR仍会携带“非人源”序列,诱导患者产生抗药物抗体
人源化和全人源抗体尽管较嵌合抗体有所改进,但仍会在临床上引发免疫应答,产生抗药物抗体(ADA)。这种免疫原性主要是因为CDR中存在T细胞表位(如下所示)。
传统的“人源化评分”无法完全反映出抗体CDR的免疫原性风险。即使识别出这样的热点,也没有成功率高的去除方法,因为CDR中的单点突变可能会使抗体失去与其靶点结合的能力。传统方法很难解决这种多目标优化问题。

人源化抗体1 (上) 和全人源抗体2 (下) 含有T细胞表位 (蓝色条块),主要位于CDR中 (绿色条块)。
参考文献:1.Spindeldreher S, Karle A, Correia E, et al. T cell epitope mapping of secukinumab and ixekizumab in healthy donors[C]//MAbs. Taylor & Francis, 2020, 12(1): 1707418.
2.Meunier S, Hamze M, Karle A, et al. Impact of human sequences in variable domains of therapeutic antibodies on the location of CD4 T-cell epitopes[J]. Cellular & Molecular Immunology, 2020, 17(6): 656-658.
-
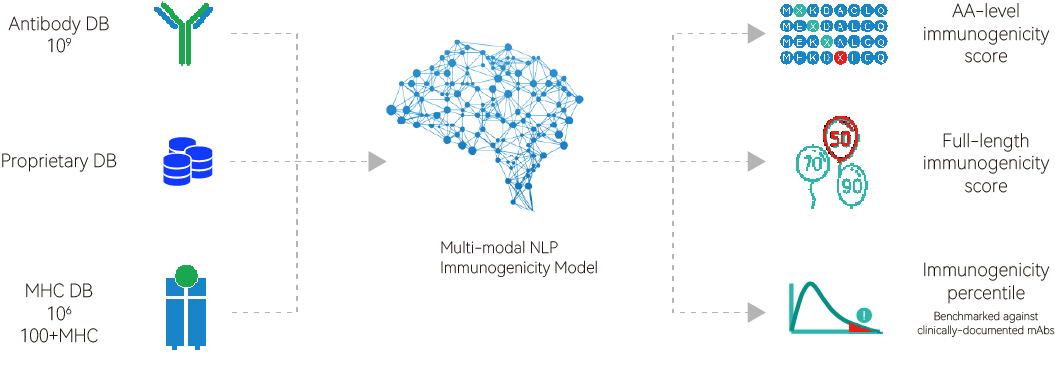
AI模型预测的免疫原性与临床数据相符
-
AI技术使我们能够更好地评估并降低免疫原性。AI模型在不同物种的大规模天然抗体序列数据库上进行了训练,可以辨别出传统算法无法检测到的细微差异。
我们开发出了多模态NLP(自然语言处理)模型,且已经过多种类型的公共和专有数据库训练。该模型可以在全序列和氨基酸水平上预测抗体的免疫原性。此外,它还可以根据临床参照抗体,对候选抗体进行百分位排序。
-
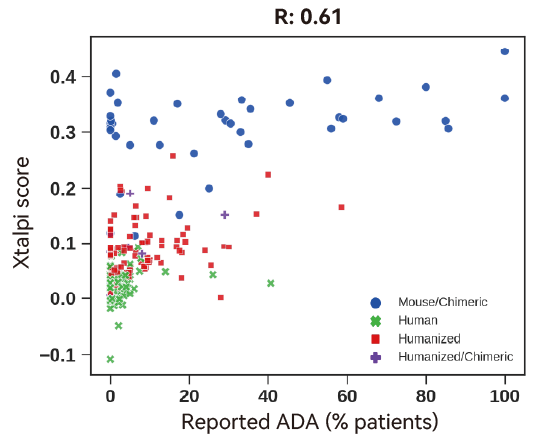
我们利用该模型预测了217种处于临床阶段且有ADA记录的抗体。结果表明,预测的ADA与实际ADA的相关性较高(R=0.61)。
该模型还可识别引起免疫原性的单个氨基酸,包括CDR中的氨基酸。(如案例1所示)
该模型构成了我们XuperHumanTM 平台的基础,该平台旨在识别并消除抗体和其他蛋白药物的免疫原性。
-
-
案例 1预测临床抗体的ADA,精确定位问题序列
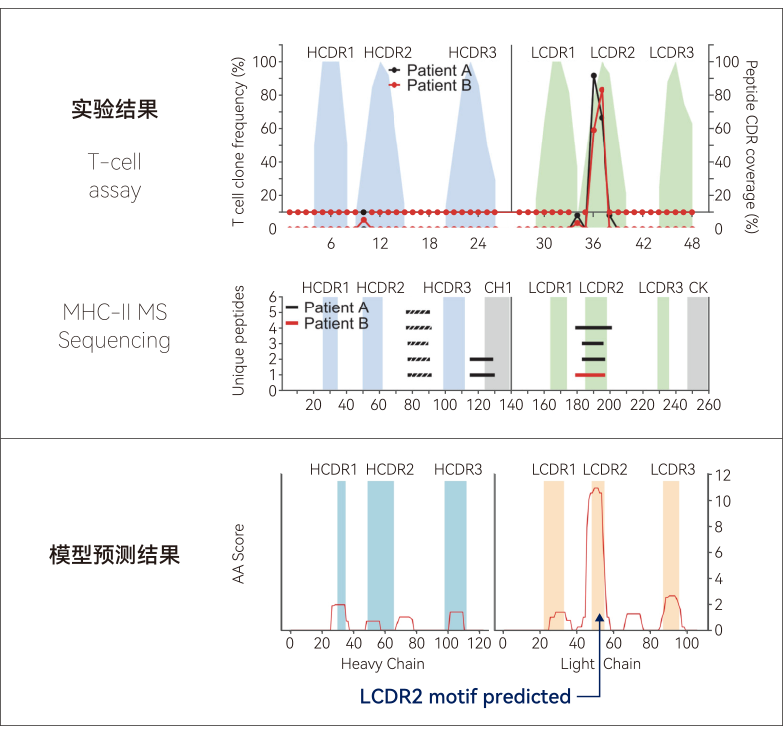
我们采用XuperHuman™AI模型预测了Natalizumab的免疫原性,这是一款已获批上市的抗体,在患者中观察到中等至较高水平的ADA。采用该模型预测的ADA发生率与临床记录相符(如下所示),而且还成功定位了此前通过体外实验识别出的潜在问题序列(如右图所示)。

-
案例 2在保持亲和力的同时去除CDR中的问题序列
-
基于之前的研究结果,我们想要去除Natalizumab的CDR中的免疫原性序列,同时保留其与靶点结合的能力。
-
在我们的计算流程中获得的前88个突变体中,有72个保持了结合能力(如下所示)。在免疫原性下降方面排名前4的突变体目前处于临床参照药物的前50%(ADA约为3%),而野生型处于最后25%(ADA约为10%)。所有4个突变体均保持了与野生型相同的结合亲和力水平。目前正在开展进一步的基于细胞实验的免疫原性测定。
-




